近日,我院高峰教授课题组通过分子动力学模拟揭示了SARS冠状病毒和新冠病毒与人类受体在不同温度下结合特性的差异,为药物设计提供了有益的指导和参考。
SARS病毒和新冠病毒对细胞的成功入侵主要取决于刺突蛋白(S蛋白)的受体结合域(RBD)与人类受体血管紧张素转化酶2(ACE2)之间的蛋白质-蛋白质相互作用(相关结构如左图所示)。
新冠疫情爆发以来,针对RBD与ACE2之间的相互作用已进行了大量的研究,但绝大多数仅在室温300 K(约27摄氏度)下进行。温度是影响病毒感染性的重要因素,此次高峰教授课题组揭示了SARS病毒和新冠病毒在不同温度(200K, 250K, 273K, 300K, 350K)下两者RBD和ACE2结合特性的差异。这项研究有助于了解SARS病毒和新冠病毒感染能力的差异。
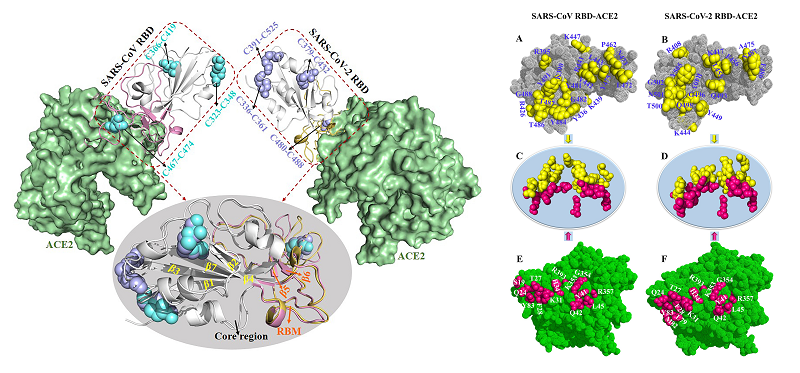
基于分子动力学模拟的平衡轨迹,高峰教授课题组发现在所选取各温度下新冠病毒RBD的均方根波动(RMSF)值都比SARS病毒低,构象分布更集中,说明新冠病毒RBD结构更为稳定。此外,SARS病毒/新冠病毒RBD与ACE2在不同温度下的结合强度也通过分子力学泊松-玻尔兹曼表面积(MM-PBSA)和溶剂化相互作用能(SIE)方法进行了评估,发现在任意所选取温度下新冠病毒RBD与ACE2的结合能力都比SARS病毒更强。随后对总结合自由能进行了残基分解,找到了RBD与ACE2结合过程中的热点残基(如右图所示),与2020年Nature报道的实验结果(Lan et al.,Nature, 2020) 高度吻合。最后还重点分析了导致SARS病毒和新冠病毒两者RBD与ACE2结合差异的关键残基,为后续相关药物设计提供了有益的指导和参考。
以上研究发表在生物信息学领域顶尖期刊《Briefings in Bioinformatics》(中科院分区一区Top期刊),论文题目“Comparison of the binding characteristics ofSARS-CoV andSARS-CoV-2 RBDs to ACE2 at different temperatures by MD simulations”(链接https://doi.org/10.1093/bib/bbab044)。我院博士生闫芳芳为该论文的第一作者,高峰教授为通讯作者。近年来,高峰教授课题组在微生物基因组生物信息学与合成生物学研究领域取得了一系列研究进展,相关成果发表于《Nucleic Acids Research》、《Briefings in Bioinformatics》、《Bioinformatics》等高水平国际刊物,获Science、Nature、Nature Biotechnology、Nature Reviews Microbiology、NatureMethods等期刊论文引用。本研究工作得到国家重点研发计划项目、国家自然科学基金创新研究群体项目、面上项目等资助支持。


